29.11.2022 - Neuer mehrschichtiger Netzwerkansatz zur Datenintegration erweist sich als enorm nützlich in der Patientenrisikostratifizierung bei Krebserkrankungen.
Artikel ist aktuell im renommierten Fachmagazin "Nucleic Acids Research" erschienen
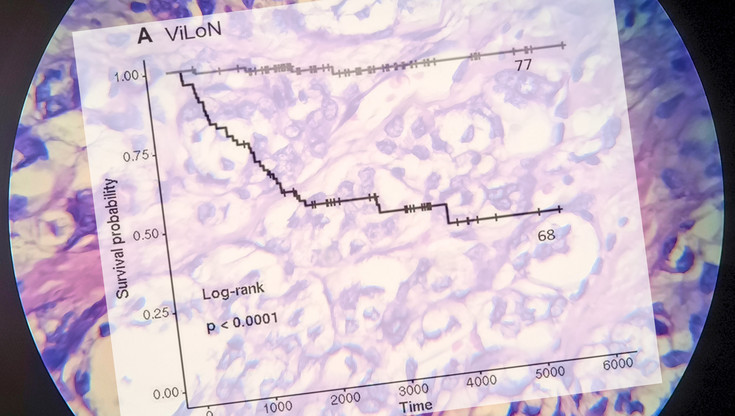
Für viele Krankheiten können Patienten ein sehr ähnliches klinisches Erscheinungsbild zeigen, obwohl die zugrundeliegenden molekularen Mechanismen für jeden sehr unterschiedlich sein können. Eine Unterscheidung von z. B. unterschiedlichen Typen von Schilddrüsenkrebs ist jedoch sowohl relevant für die klinische Behandlung als auch das Verständnis der Krankheit und die Entwicklung neuer Therapien.
Einem Forschungs-Team am Institut für Molekulare Biotechnologie an der BOKU unter der Leitung von David Kreil ist es jetzt gelungen, einen Algorithmus mit einem neuartigen, netzwerkbasierten Ansatz für die Integration unterschiedlicher molekularer Profile zu entwickeln: „Dieser Algorithmus ermöglich uns, Patient*innen, deren Krankheit einen ähnlichen zugrundeliegenden Mechanismus haben, zusammen zu gruppieren.“
Mehrschichtiger Netzwerkansatz zur Datenintegration
Dank der immer umfangreicheren biomedizinischen Datensammlung machen sich moderne netzwerkbasierte Darstellungen die Komplementarität der verschiedenen Datenquellen sowie die Ähnlichkeiten zwischen verschiedenen Patienten zunutze. „Neu an unserem Algorithmus „Variation of information fused Layers of Networks“ – kurz: ViLoN – ist, dass er (a) verschiedene Datentypen, (b) den Blick auf die Prozessebene und (c) die gemeinsame Analyse verschiedener Patient*innen zusammenbringt. Mit Hilfe dessen kann dann über die sensitive Zusammengruppierung eine gemeinsame Diagnose und ähnliche Prognosen erstellen“, betont Kreil.
Das BOKU-Team hat ViLoN an sechs verschiedenen Krebsarten, darunter durchaus diagnostisch herausfordernde Typen, validiert und diese mit dem besonderen Fokus auf die Überlebenswahrscheinlichkeit untersucht. Praktischer Benefit für den Patienten/die Patientin: Die Identifizierung, ob es sich z. B. um einen aggressiven oder einen möglichst gutartigen Krebs handelt oder welche molekularen Mechanismen dem Krebs zugrundeliegen, führt zu einer besseren Risikovorhersage und erleichtert in Folge die Entscheidung für die Auswahl der Therapie. „Eine weitere Schlüssel-Innovation unseres ViLoN-Ansatzes besteht darin, dass er auch für kleinere Patientenkohorten funktioniert – und ebenso für Krankheiten, für die es bisher nur sehr schwierig oder noch gar nicht möglich war“, so Kreil.
In ihrem Artikel in der aktuellen Ausgabe des Fachmagazin Nucleic Acids Research haben Daniel Kreil und sein Team die Analyse, welche molekulare Mechanismen besonders gutmütigen oder bösartigen Untertypen einer Krebsart zugrunde liegen, für Schilddrüsenkrebs und Neuroblastoma durchgeführt. Auch im Bereich der Grundlagenforschung leistet ViLoN so hervorragende Dienste, da er vom Forscher*innen-Team an sehr unterschiedlichen Datensätzen getestet wurde und somit für andere Forscher*innen und Situationen gut einsetzbar ist. „Somit öffnet unser Algorithmus auch Türen für die Erforschung unterschiedlichster molekularer Mechanismen und neuer Therapien“, freut sich Kreil.
Artikel in Nucleic Acids Research
https://academic.oup.com/nar/advance-article-abstract/doi/10.1093/nar/gkac988/6827105
Über die Forschungs-Gruppe an der BOKU
Die Forschungsgruppe von David Kreil wurde 2005 über eine WWTF Stiftungsprofessur an der BOKU gegründet, 2008 dauerhaft etabliert und seit 2019 in das neu etablierte Instituts für Molekulare Biotechnologie eingegliedert. Sie arbeitet an der Schnittstelle zwischen Experiment und Computeranalyse und setzt in enger Kooperation mit der US-Regulierungsbehörde für Medikamente FDA weltweit Standards in der Durchführung und Analyse in der Gentranskript-Analyse und Hochdurchsatz-Sequenzierung (Next-Generation Sequencing).
Über Gruppenleiter David Kreil
Ausgebildet an der TU München, der Universität Cambridge, und dem Europäischen Bioinformatik Institut EMBL-EBI kam David Kreil 2005 über eine WWTF Stiftungsprofessor an die BOKU Wien, und leitet dort seither eine kleine hocheffektive Forschungsgruppe, deren Alumni sich in leitenden Rollen in der biomedizinischen Industrie und führenden Forschungsinstituten finden. Seit 2019 ist er auch wissenschaftlicher Direktor des Institute for Advanced Research in Artificial Intelligence IARAI (www.iarai.ac.at).
Kontakt
Assoc.Prof. Dr. David Kreil
Universität für Bodenkultur Wien
Institut für Molekulare Biotechnologie
Email: d.kreil(at)boku.ac.at
Telefon: 0660 9899903
